高通量测序
高通量测序
一、实验流程
16S/18S/ITS rDNA,序列包括保守区域和高变区域,其中保守区在微生物菌种间差异不
大,高变区具有属或种的特异性,随亲缘关系不同而有一定的差异。因此,16S/18S/ITS
rDNA 可以做作为揭示生物物种的特征核酸序列,被认为是适于微生物系统发育和分类鉴定
的指标。如今,16S/18S/ITS rDNA 扩增子测序技术已成为研究环境样本中微生物群落组成
结构的重要手段。
16S rDNA 扩增子测序,是通过特异性引物扩增样本中原核生物16S rDNA 的可变区,
构建高通量测序文库并对16S rDNA 可变区序列进行分析,从而鉴定环境中原核微生物的组
成与丰度的方法。GENEWIZ 自主研发的引物体系能有效扩增出16S rDNA 的多个可变区(V3,
V4),能够准确鉴定出包含古生菌在内的多个物种。18S/ITS rDNA 扩增子测序,是通过特
异性引物扩增样本中真核生物18S/ITS rDNA 的可变区,构建高通量测序文库并对18S/ITS
rDNA 可变区序列进行分析,从而鉴定环境中真核微生物的组成与丰度的方法。Illumina
MiSeq 测序平台由于在测序深度、通量、运行周期、测序准确性及价格方面的优势,广泛应
用于16S/18S/ITS rDNA 扩增子测序;近年来,Paired-end Reads 拼接方法使MiSeq 测序平台
的读长达到了600bp,从而使分析准确性得到进一步提高。
16S/18S/ITS rDNA 扩增子测序的实验流程包括样本基因组DNA 的提取与质检,
16S/18S/ITS rDNA 可变区扩增与测序文库构建,高通量测序等多个步骤。其中每一个环节
都会对数据质量和数量产生影响,而数据质量又会直接影响后续信息分析的结果,为了保证
源头数据的准确性与可靠性,我们对每一步实验过程都进行严格质控,检测合格后,按照目
标数据量调整文库体积,将多个文库混合后进行Illumina MiSeq 测序。具体实验流程如下:

Figure 一.1 菌群多样性测序实验流程
二、生物信息分析流程
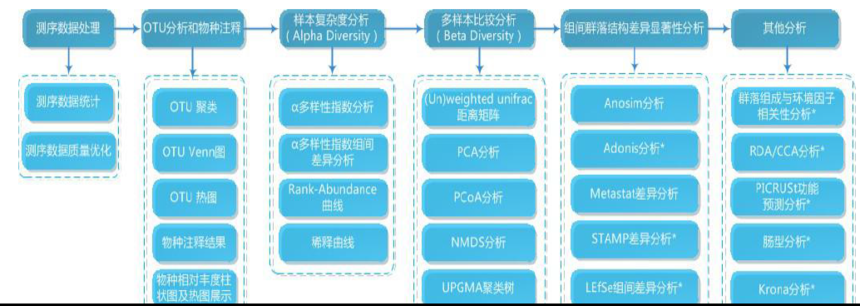
首先对原始数据进行去接头和低质量过滤处理,然后去除嵌合体序列,得到有效序列后
进行聚类分析,每一个聚类称为一个物种操作单元(Operational Taxonomic Units,OTU), 对
OTU 的代表序列作分类学分析,得到各样本的物种分布信息。基于OTU 分析结果,可以对
各个样本进行多种α多样性指数分析,得到各样本物种丰富度和均匀度信息等;基于分类学
信息,可以在各个分类水平上进行群落结构的统计分析;通过计算Unifrac 距离、构建UPGMA
样本聚类树、绘制PCoA 等图,可以直观展示不同样本或分组之间群落结构差异。
在上述分析的基础上,还可以进行一系列深入挖掘,如通过多种统计分析方法分析分组
样本间的群落结构差异性,结合环境因子和物种多样性关联分析,挖掘显著影响组间群落结
构变化的环境影响因子等。
二、生物信息分析流程
首先对原始数据进行去接头和低质量过滤处理,然后去除嵌合体序列,得到有效序列后
进行聚类分析,每一个聚类称为一个物种操作单元(Operational Taxonomic Units,OTU), 对
OTU 的代表序列作分类学分析,得到各样本的物种分布信息。基于OTU 分析结果,可以对
各个样本进行多种α多样性指数分析,得到各样本物种丰富度和均匀度信息等;基于分类学
信息,可以在各个分类水平上进行群落结构的统计分析;通过计算Unifrac 距离、构建UPGMA
样本聚类树、绘制PCoA 等图,可以直观展示不同样本或分组之间群落结构差异。
在上述分析的基础上,还可以进行一系列深入挖掘,如通过多种统计分析方法分析分组
样本间的群落结构差异性,结合环境因子和物种多样性关联分析,挖掘显著影响组间群落结
构变化的环境影响因子等。
推荐
-

-

QQ空间
-

新浪微博
-

人人网
-

豆瓣

